Bezpłatną platformę internetową o degronach - komórkowych znacznikach, dzięki którym rozpoznawane są białka przeznaczone do degradacji - udostępnił zespół badaczy z Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie (MIBMiK). “To cenne narzędzie dla naukowców zajmujących się biochemią, biologią molekularną oraz medycyną” - komentują przedstawiciele instytucji.
Zespół dr. hab. Wojciecha Pokrzywy opracował zaawansowane narzędzie do wieloaspektowej analizy degronów, które zostało opublikowane w prestiżowym Nucleic Acids Research.
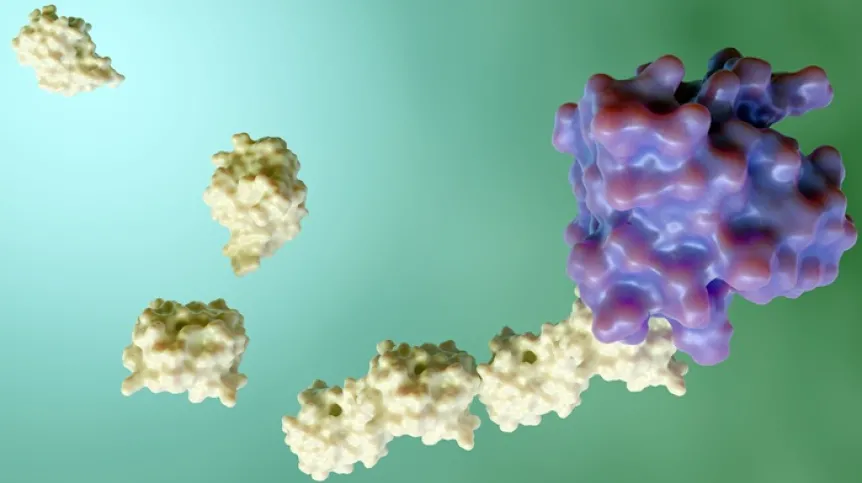
Degrony to krótkie fragmenty białek odgrywające kluczową rolę w regulacji procesu niszczenia niepotrzebnych już w komórce białek. Degrony są rozpoznawane i wiązane przez pewne ważne enzymy szlaku degradacyjnego - ligazy ubikwityny E3. Jeśli zaś enzym ten przyłączy do danego białka (które zawiera wspomniany degron) kilka cząsteczek ubikwityny, oznacza to dla białka wyrok śmierci - białko zostanie zniszczone przez proteasom, czyli komórkowe centrum recyklingu, a uwolnione w tym procesie aminokwasy będą mogły posłużyć za budulec nowych białek. W ten sposób degrony są niezbędne w procesie "czyszczenia" komórki ze zbędnych lub wadliwych białek, co zapewnia komórce prawidłowe funkcjonowanie.
Mimo intensywnych badań nad funkcjonowaniem degronów, do tej pory brakowało zasobów umożliwiających ich systematyczne i wielopłaszczyznowe badania.
DEGRONOPEDIA została stworzona, aby uporządkować wiedzę o degronach. Umożliwia przeszukiwanie proteomu — pełnego katalogu białek w komórce wybranego gatunku — pod kątem obecności degronów i mapowania ich w kontekście ubikwitynacji. Platforma ta udostępnia również informacje o nieuporządkowanych obszarach białek, które mogą odgrywać istotną rolę podczas ich degradacji. Ponadto narzędzie to dostarcza danych na temat ewolucyjnej konserwacji degronów, ich modyfikacji oraz mutacji.
"DEGRONOPEDIA wykorzystuje technologie uczenia maszynowego w celu identyfikacji degronów, na przykład w nowo powstałych końcach białkowych podczas proteolizy, czyli pękania sekwencji białkowych. Praktyczne zastosowanie platformy oraz wyników modelu uczenia maszynowego zostało potwierdzone badaniami eksperymentalnymi" — mówi współautorka artykułu i liderka projektu, doktorantka Natalia Szulc z Laboratorium Metabolizmu Białek MIBMiK, cytowana w komunikacie swojego instytutu.
DEGRONOPEDIA znajduje się na stronie internetowej. Są tam dostępne dane o poznanych dotąd degronach, głównie ludzkich oraz drożdżowych - na razie to ponad 1800 motywów.
"DEGRONOPEDIA jest cały czas rozwijana, wraz z pojawieniem się nowych badań, regularnie dodajemy nowe degrony. Ponadto, mamy plany na dalszy rozwój naszej platformy, obejmujące wprowadzenie nowych funkcjonalności, w tym wytrenowanie dodatkowych modeli uczenia maszynowego" – informuje PAP dr hab. Wojciech Pokrzywa.
O tym, jak działa cały proces degradacji białek, w tym degrony, można dowiedzieć się dzięki darmowej interaktywnej grze komputerowej DEGRADATOR, którą również opracowali dr. hab. Wojciech Pokrzywa i Natalia Szulc. W tej grze gracz wciela się w postać ligazy ubikwityny E3 i dołącza łańcuchy ubikwityny do białek, co prowadzi do ich zniszczenia przez proteasom. DEGRADATOR przedstawia również przełomową technologię celowanej degradacji białek na przykładzie związków PROTAC, które mogą wkrótce zrewolucjonizować terapie chorób nowotworowych bądź neurodegeneracyjnych. Gra dostępna jest bezpłatnie na tej stronie i w Google Play.
PAP - Nauka w Polsce, Ludwika Tomala
lt/ zan/
Fundacja PAP zezwala na bezpłatny przedruk artykułów z Serwisu Nauka w Polsce pod warunkiem mailowego poinformowania nas raz w miesiącu o fakcie korzystania z serwisu oraz podania źródła artykułu. W portalach i serwisach internetowych prosimy o zamieszczenie podlinkowanego adresu: Źródło: naukawpolsce.pl, a w czasopismach adnotacji: Źródło: Serwis Nauka w Polsce - naukawpolsce.pl. Powyższe zezwolenie nie dotyczy: informacji z kategorii "Świat" oraz wszelkich fotografii i materiałów wideo.