
W próbkach ludzkich odchodów sprzed ponad tysiąca lat i we wnętrznościach starożytnej mumii znaleziono dobrze zachowane DNA kilkuset gatunków ówczesnych bakteriofagów - wirusów infekujących bakterie. Okazało się, że genom jednego z gatunków jest niemal taki sam, jak u współczesnych wirusów. Dlaczego ewolucja tego gatunku stanęła w miejscu na tak długo?
Wyniki badań międzynarodowego zespołu, kierowanego przez Polaków ukazały się w “Nature Communications” i zmieniają spojrzenie na “wyścig zbrojeń” między wirusami a ich bakteryjnymi gospodarzami.
“To, co znaleźliśmy, wydaje się sprzeczne z tym, co dotąd było wiadomo o bakteriofagach” - komentuje w rozmowie z PAP pierwszy autor badań Piotr Rozwalak z Uniwersytetu im. Adama Mickiewicza w Poznaniu (UAM). Badania te przeprowadzone były w ramach jego pracy magisterskiej.
Wirusy, aby się namnażać, muszą dostać się do komórki gospodarza i sprawić, by skopiowała ona jego materiał genetyczny. Są wirusy, które atakują komórki roślin, ludzi czy zwierząt. Infekcje wirusowe grożą również komórkom bakterii. Takie specyficzne dla bakterii wirusy to bakteriofagi (czy też krócej: fagi).
Zarówno bakterie, jak i bakteriofagi zamieszkują ludzki układ pokarmowy. Niektóre sprzyjają zdrowiu człowieka lub są neutralne, a inne - niekoniecznie. Ich DNA znaleźć można w próbkach odchodów czy materiale pobranym z jelit.
Bioinformatyk i współautor artykułu, dr Andrzej Zieleziński z UAM, tłumaczy, że dzięki zdobyczom metagenomiki można sekwencjonować i odczytywać fragmenty DNA znajdujące się w próbce materiału biologicznego pobranego z dowolnego środowiska (np. gleby czy układu pokarmowego człowieka). Następnie można składać te pocięte “puzzle” w większe fragmenty, w tym również całe genomy i porównywać je z już znanymi genomami różnych organizmów.
Dzięki temu można np. zsekwencjonować miliony fragmentów nici DNA zawarte w jakiejś próbce materiału, nawet jeśli próbka ta liczy tysiące czy miliony lat, ale jest świetnie zachowana.
RECYKLING PALEOODCHODÓW
W ten sposób, zespoły z całego świata, poddawały badaniom ludzkie odchody sprzed ponad tysiąca lat zachowane w pustynnych jaskiniach Meksyku, Arizony czy Utah w Stanach Zjednoczonych. Próbki pobrano także z wnętrzności ludzkiej mumii sprzed 5000 lat znalezionej w Alpach, czy w kopalni soli sięgającej epoki brązu.
Co ciekawe dane z tych badań były publicznie dostępne. Żeby uzyskać dostęp do danych o kopalnym DNA zawartym w paleoodchodach, nie trzeba więc było przeprowadzać kosztownych poszukiwań, badań archeologicznych i sekwencjonować genomów, wystarczyło tylko przeanalizować publicznie dostępne bazy danych i poszukać w nich innych informacji niż szukali na początku ich autorzy.
“My właśnie przeprowadziliśmy taki recykling danych” - tłumaczy Piotr Rozwalak.
O ile wcześniejsze badania innych zespołów skupiały się na bakteriach flory jelitowej sprzed wieków, o tyle zespół z udziałem Polaków postanowił się skupić na genomie pradawnych bakteriofagów zamieszkujących ludzki układ pokarmowy. "W ten sposób postawiliśmy krok na polu nowej dziedziny wiedzy, jaką jest wiromika, czyli nauka o wirusach obecnych w różnych próbkach środowiskowych. Jako jedni z pierwszych, przebadaliśmy próbki pod kątem bakteriofagów. I otworzyliśmy tym samym drzwi to zupełnie nowej poddziedziny, jaką jest kopalna wiromika, zajmująca się badaniem wirusów z przeszłości ” - tłumaczy Piotr Rozwalak.
WYŚCIG ZBROJEŃ
Naukowcy postanowili sprawdzić, jak szybko zmieniał się przez ostatnie stulecia genom bakteryjnych wirusów. Zakładali, że przynajmniej niektóre kopalne bakteriofagi będą podobne do tych współczesnych. Zdawali sobie jednak sprawę, że przeszkodą może być tu ewolucyjny proces, który zaciera ślady podobieństwa między kolejnymi pokoleniami wirusów. Chodzi o ciągły wyścig zbrojeń między bakteriami i wirusami, który może w miarę szybko zmienić obie strony nie do poznania.
Bakteriofagi przyczyniają się często do śmierci lub osłabienia bakterii. W interesie bakterii jest więc zwykle to, by wytwarzać skuteczne mechanizmy obrony przed swoimi patogenami. Jeśli jedna bakteria znajdzie sposób, by obronić się przed infekcją, może przekazać taką genetyczną "aktualizację oprogramowania" swoim sąsiadom. A wtedy namnażać się będą tylko te wirusy, które znajdą nową wyrwę w zabezpieczeniach gospodarza. Wydawało się więc, że bakteriofagi i ich gospodarze zawsze ewoluują bardzo szybko.
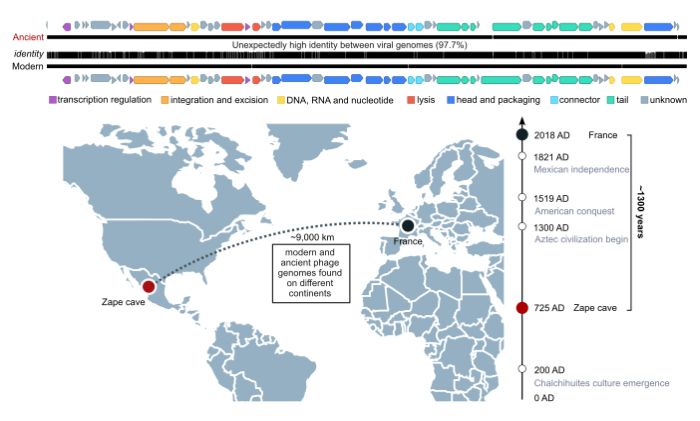
A tu niespodzianka. Badacze znaleźli genomy niemal 300 gatunków bakteriofagów, które częściowo były podobne do tych obecnych. Jeden z wirusów był jednak wyjątkowy: jego sekwencja genomowa była aż w 97,7 procentach podobna do genomu współczesnego bakteriofaga. Chodzi o wirusa o nazwie Mushuvirus mushu. To wirus zakażający beztlenowe bakterie jelitowe, które mają pozytywny wpływ na nasze zdrowie, w tym z rodzaju Faecalibacterium.
NIEZMIENNIE
Co sprawiło, że potomkowie wirusa wyglądają tak samo, jak ich przodkowie sprzed 1300 lat? “To wskazuje na długoterminową relację między tym wirusem a jego gospodarzem” - piszą autorzy publikacji.
Piotr Rozwalak opowiada: “Nasz wirus wbudowuje się w genom bakterii. A bakteria z jakiegoś powodu nie broni się przed tym. Naszą hipotezą jest to, że dochodzi tam do mutualistycznej relacji: każda ze stron korzysta jakoś z obecności drugiej strony - coś otrzymuje i chce kooperować. Możemy więc na podstawie tych badań domyślać się, że czasem bakterie mogą współpracować z fagami i mieć jakiś zysk z ich obecności”.
Dr Zieleziński dodatkowo sugeruje, że stabilność wirusa może również wynikać z jego zdolności do infekowania aż 8 różnych gatunków bakterii. A to świadczy w świecie wirusów o dużej wszechstronności. W takiej jednak sytuacji znaczne zmiany w DNA wirusa mogą prowadzić do utraty jego zdolności do infekowania któregoś z gospodarzy. Wirus nie ma więc interesu w tym, żeby ewoluować.
PODCHODY W ODCHODACH
Dr Zieleziński wyjaśnia, że w badaniach nad kopalnym DNA należy być wyjątkowo sceptycznym, ponieważ istnieje możliwość przypadkowego zanieczyszczenia próbek współczesnym DNA. Dlatego autorzy przeprowadzili serię rygorystycznych testów mających na celu weryfikację autentyczności kopalnego wirusa. Wyniki testów jednoznacznie pokazały, że Mushuvirus mushu był równie stary jak badana próbka.
“Dokonaliśmy pierwszej na świecie rekonstrukcji genomów kopalnych bakteriofagów w próbkach reprezentujących środowisko ludzkich jelit sprzed nawet 5000 lat i opracowaliśmy katalog tych wirusów, rzucając nieco światła na nieznane wcześniej aspekty historii mikroświata jelitowego. A przy okazji odkryliśmy jeden gatunek wirusa, który nie tylko przetrwał przez wieki w jelicie człowieka, ale również zachował swoją niezmienioną formę od co najmniej 1300 lat.” - podsumowuje badania dr Zieleziński.
Nauka w Polsce, Ludwika Tomala
lt/ agt/
Fundacja PAP zezwala na bezpłatny przedruk artykułów z Serwisu Nauka w Polsce pod warunkiem mailowego poinformowania nas raz w miesiącu o fakcie korzystania z serwisu oraz podania źródła artykułu. W portalach i serwisach internetowych prosimy o zamieszczenie podlinkowanego adresu: Źródło: naukawpolsce.pl, a w czasopismach adnotacji: Źródło: Serwis Nauka w Polsce - naukawpolsce.pl. Powyższe zezwolenie nie dotyczy: informacji z kategorii "Świat" oraz wszelkich fotografii i materiałów wideo.













